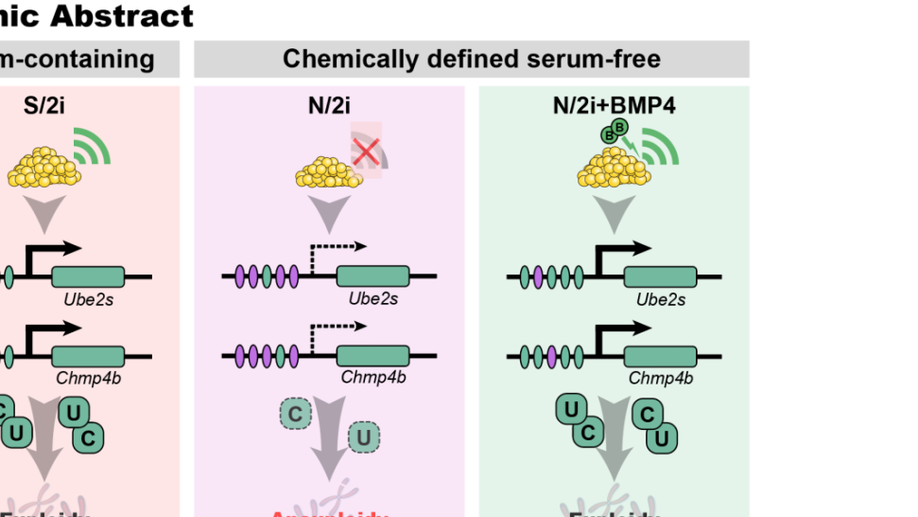
BMP4可调控下游靶基因Ube2s和Chmp4b来维持小鼠胚胎干细胞发育潜能
2022年2月11日,同济大学生命科学与技术学院高绍荣、陈嘉瑜和江赐忠课题组于Protein & Cell 在线发表了题为BMP4 preserves the developmental potential of mESCs through Ube2s- and Chmp4b- mediated chromosomal stability safeguarding 的研究论文。这项工作揭示了骨形态发生蛋白BMP4对于小鼠胚胎干细胞多能性的维持有显著的促进作用,在无血清培养体系中添加BMP4 可以适当激活 BMP 信号通路,进而上调无血清体系中受损的下游靶基因Ube2s和Chmp4b表达,来保障小鼠胚胎干细胞的染色体完整性和增殖能力,并进一步提高并长期维持小鼠胚胎干细胞的体内发育潜能。
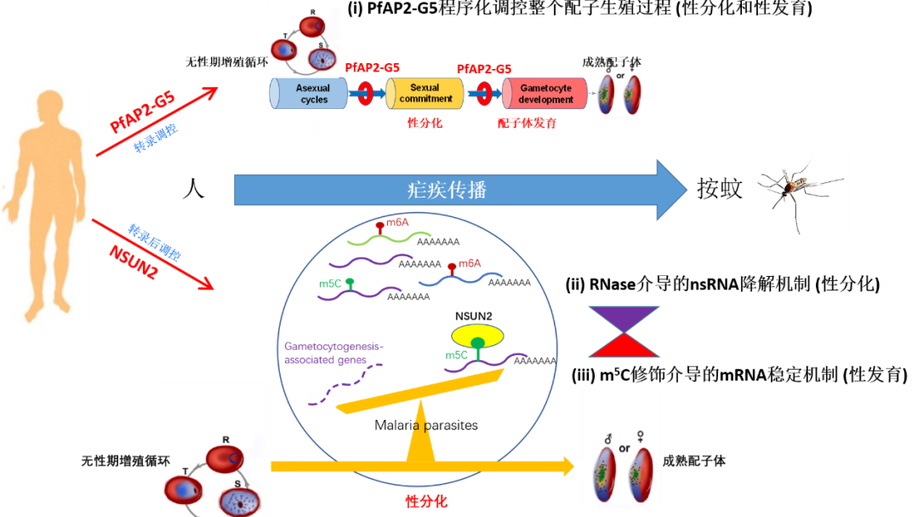
揭示表观转录组调控疟疾传播的新机制
2022年2月25日,同济大学医学院张青锋课题组联合生科院江赐忠、厦门大学袁晶及美国国立卫生院Thomas Wellems等团队在美国科学院院刊PNAS上发表了题为5-methylcytosine modification by Plasmodium NSUN2 stabilizes mRNA and mediates the development of gametocytes的研究性论著,首次揭示了表观转录组调控疟原虫配子生殖的表观遗传调控新机制,鉴定了新调控因子NSUN2并阐明其作用机制,为疟疾传播阻断新措施的研发提供了新靶点

疟疾传播的核心调控因子
2021年8月8日,同济大学医学院/附属东方医院张青锋团队联合生命科学院江赐忠团队及江苏省寄生虫病防治研究所曹俊团队在Nucleic Acids Research杂志在线发表了题为A cascade of transcriptional repression determines sexual commitment and development in Plasmodium falciparum的研究性论著,首次揭示了恶性疟原虫配子生殖的多重表观遗传调控网络,发现了核心调控因子PfAP2-G5并阐明其作用机制,为疟疾传播阻断新措施的研发提供了关键靶点。
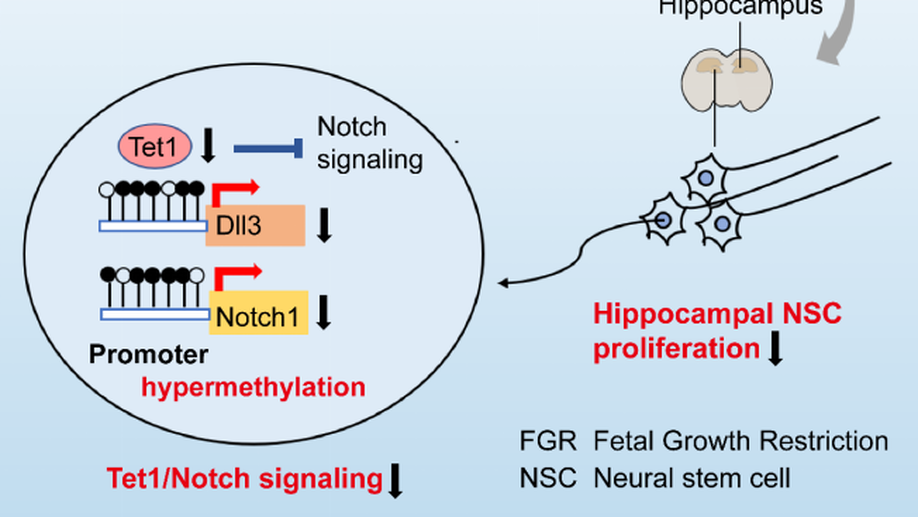
Cell Reports:揭示胎儿生长受限子代认知障碍新机制
2021 年 11 月 2 日,同济大学附属第一妇婴保健院康九红教授课题组在《Cell Reports》杂志在线发表题为 Fetal Growth Restriction Impairs Hippocampal Neurogenesis and Cognition via Tet1 in Offspring 的研究论文,该研究揭示了 DNA 羟甲基化酶 Tet1 调控宫内生长受限子代海马神经发生受损和认知缺陷的作用和机制。

新型体细胞核移植挽救方法的建立及表观重编程机制对比研究
2020年10月12日,同济大学生命科学与技术学院高绍荣团队与江赐忠团队在《Cell Stem Cell》 杂志在线发表了题为“Parallel analysis of H3K9ac reveals Dux-dependent 2-cell genome activation deficiency in somatic cell nuclear transfer embryos”的研究成果。他们首先通过对于早期正常受精小鼠,核移植小鼠,以及TSA挽救处理后的核移植小鼠的H3K9ac组蛋白修饰的平行分析,鉴定出一系列以H3K9ac标记的重编程异常区域(aberrantly acetylated regions, AARs),并首次揭示出TSA挽救的原有表观环境依赖性(歧视性挽救)特性。接着,作者通过TF footprint分析,利用已建立的H3K9ac图谱推断出潜在的转录因子挽救靶点Dux,成功进行了核移植胚胎挽救并进行了后续深入的机制论证。
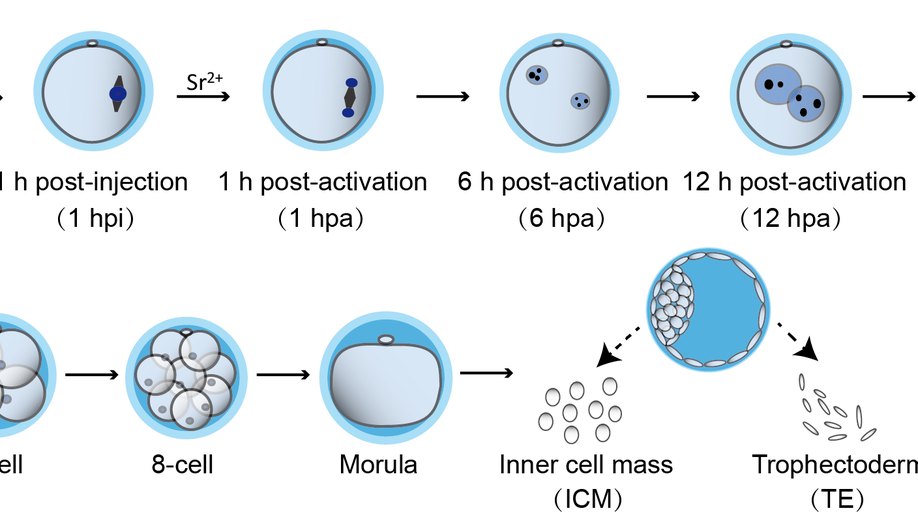
小鼠体细胞核移植过程中染色质高级结构重编程模式及分子机制
2020年4月14日,同济大学生命科学与技术学院高绍荣团队与江赐忠团队在《Nature Communications》 杂志在线发表了题为“Chromatin architecture reorganization in murine somatic cell nuclear transfer embryos”的研究成果。他们采用了经过优化的少量细胞全基因组染色质构象捕获技术(sisHi-C),对小鼠SCNT胚胎发育过程进行连续采样,并详细描绘了SCNT植入前胚胎染色质高级结构的动态变化过程。
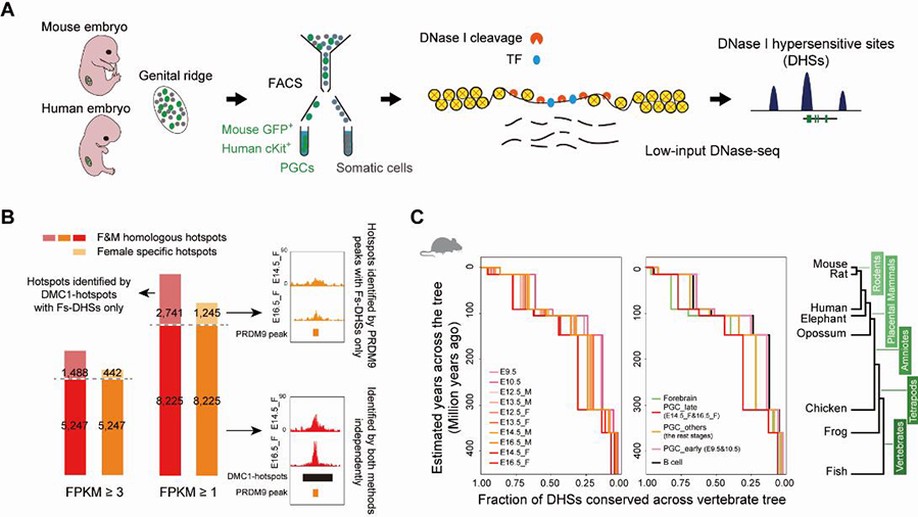
高绍荣/江赐忠教授团队在Cell Research在线发表论文揭示原始生殖细胞的染色质状态
10月10日,同济大学高绍荣课题组联合江赐忠课题组在Cell Research杂志在线发表了题为“Accurate annotation of accessible chromatin in mouse and human primordial germ cells”的最新研究成果,他们利用最新优化的low-input DNase-seq技术结合生物信息学大数据分析,精确注释了PGCs发育过程中染色质的开放状态,为PGCs发育过程中各个重要生物学事件提供了重要线索。
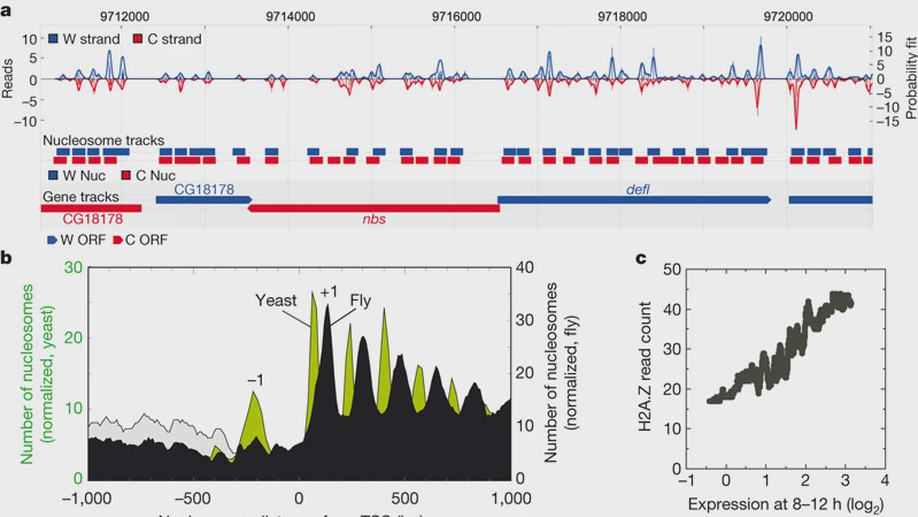
Nucleosome organization in the Drosophila genome
Comparative genomics of nucleosome positions provides a powerful means for understanding how the organization of chromatin and the transcription machinery co-evolve. Here we produce a high-resolution reference map of H2A.Z and bulk nucleosome locations across the genome of the fly Drosophila melanogaster and compare it to that from the yeast Saccharomyces cerevisiae. Like Saccharomyces, Drosophila nucleosomes are organized around active transcription start sites in a canonical -1, nucleosome-free region, +1 arrangement. However, Drosophila does not incorporate H2A.Z into the -1 nucleosome and does not bury its transcriptional start site in the +1 nucleosome. At thousands of genes, RNA polymerase II engages the +1 nucleosome and pauses. How the transcription initiation machinery contends with the +1 nucleosome seems to be fundamentally different across major eukaryotic lines.